DNA dizilimi, laboratuvarlarda kullanılan önemli bir teknik olup DNA’da yer alan baz sırasının belirlenmesidir. DNA dizilimi için çok çeşitli yöntemler kullanılmaktadır. Bu yöntemler temelde klasik ve yeni nesil dizileme olarak sınıflandırılmaktadır.
Klasik yöntemlere, Sanger ve Maxim Gilbert yöntemleri örnek verilebilir. Klasik yöntemler ile dizilemenin çok zaman alması, uğraştırıcı olması, maliyetli olması ayrıca bilimsel çalışma sayısının artması gibi sebeplerden yeni yöntemlere ihtiyaç duyulmuştur. Teknolojinin gelişmesi ile de yeni nesil dizilimler keşfedilmiştir. Yeni nesil dizileme teknolojilerine Illumina, Ion Torrent, Roche, PacBio Real Time Sequencing, Helicos ve Oxford Nanopore örnek verilebilir. Yeni nesil genom dizileme teknolojileri ile geniş kapsamlı analizler daha kısa sürede ve maliyette gerçekleştirilmektedir. Pratik ve kolay uygulanabilir olması açısından yeni nesil dizileme, birçok hastalığın tanı ve tedavisinde kullanılmaktadır. Yeni nesil gen düzenlemeyle baz ya da gen dizisinde herhangi bir değişiklik olması, mutasyon meydana gelmesi ya da olması gerekenden farklı bir durum gerçekleşmesi gözlemlenebilmektedir.
Hastalığın genetik temelini tespit edince tedavi için DNA’da belirli değişiklikler ya da düzenlemeler yapılmasına “genom düzenleme” denir. Canlı hücrelerin genomlarında spesifik DNA dizilerinin hedeflenen modifikasyonunu kolaylaştırmak için tasarlanan genom düzenleme, hücre içindeki DNA’nın hatasız ve verimli şekilde modifiye edilmesi için kullanılan bir tekniktir. Bu teknolojilerin yaygın olarak kullanılması ve araştırma konusu haline gelmesinde, özelleştirilebilme kolaylığı ve çoklu hücre tiplerinde uygulanması etkili olmuştur. Genom düzenlemede bir dizi spesifik nükleazlar kullanılır. Bu nükleazlar DNA kesim (restriction) enzimlerine benzer şekilde, genomda düzenleme yapılmak istenen bölgede DNA’yı çift iplikli olarak keser. Oluşan bu çift iplik kesikleri, hücrenin DNA tamir mekanizmaları olan Homolog Rekombinasyon (Homologous Recombination – HR) ya da Homolog Olmayan Uçların Birleşmesi (Non-Homologous End Joining – NHEJ) yoluyla tamir edilirken, hücre tarafından hangi tamir mekanizmasının kullanıldığına bağlı olarak genomda farklı modifikasyonlar meydana gelir.
Dizi spesifik nükleazlar; Çinko Parmak Nükleazları (Zinc Finger Nucleases – ZNF), Yazım Etkinleştirici Benzeri Etkileyici Nükleazlar (Transcription activator-like effector nucleases – TALENs), Kümelenmiş Düzenli Aralıklı Kısa Palindromik Yinelemeler ( Clustered Regularly Interspaced Short Palindromic Repeats – CRISPR) ve meganükleazlardır.
Genom hedefleme teknikleri -belirli bir dizinin başka yerlerde gen kopyalarının yerleştirilmesinden ziyade normal genomik konumdaki hedefe yönelik modifikasyon- ratlarda germline mühendisliği için 1980’lerde öncü olmuştur. Bu ilk çalışmalar, geleneksel gen transfer metotlarına göre gen hedeflemenin bilimsel avantajları üzerine birçok araştırmayı ve düşünceyi geliştirmiştir. ZFN, TALENs ve meganükleazlar 2010 yılına kadar süren çalışmaların geliştirilmesiyle sonuçlanmıştır. Memeli hücrelerinde RNA güdümlü bir nükleazın özellikle hedef dizileri ayırdığı ve kesin genom modifikasyonu için yeni bir yaklaşım sağladığı 2013 yılı başlarında gösterilmiştir. Günümüze kadar diğer bakteri türlerinden ilave RNA güdümlü nükleazlar tanımlanmış ve genom düzenleme olarak potansiyelleri araştırılmaktadır.
ZFN
Tek tek ele almak gerekirse, ZFN’ler kullanıcının genomda modifiye etmek istediği bölgeye göre dizayn edilen ilk dizi spesifik nükleazlardır. ZNF etkili, hedeflenmiş nükleazların ilk aşamasını temsil eder. ZFN serileri ZFN oluşumu için spesifik gen lokusuna bağlanmayı tasarlar ve daha sonra fokl nükleazıyla kaynaştırılır. Komşu iki bölgeyi tanıyan eşleştirilmiş ZFN’ler DNA’yı ikiye böler ve HDR başlatılır. Uzun sentez süresi ve modüler olmayan birleşim işlemi nedeniyle ZFN’lerin kullanımı sınırlandırılır. Bilişim aletleri hedefleme mekanizmasının gelişimine yardımcı olmasına rağmen her genomik lokusa uygun ZFN çiftleri tasarlamak mümkün değildir.
TALENs
Dizi spesifik nükleaz ailesinin ikinci üyesi olan TALEN sistemi ZFN sistemine bir alternatif olarak ortaya çıkmıştır. İlk kez 2011 yılında rapor edilen TALEN’ler genom mühendisliğinde büyük çığır açmıştır. Bu modüler sistem fokl endonükleazıyla kaynaştırılan Xanthomonas spp’den izole edilmiş TAL etkileyici DNA bağlayıcı proteinlerden temel alır. Addgene’nin en gözde kiti haline gelen Golden Gate TALEN kitiyle birlikte TALEN teknolojisi, araştırma topluluğu tarafından hızla benimsenmiştir. TALEN’lerin ihtiyaca göre düzenlenebilen DNA bağlama özellikleri ayrıca gen aktarımının ayarlanması için özel transkripsiyon faktörlerinin tasarımına imkan sağlamıştır.
CRISPR
CRISPR, kullanıma başlanmasıyla genom mühendisliği çalışmaları oldukça hızlanmış olup bitki biyoteknolojisinde de çığır açmıştır. CRISPR’ın çalışma prensibi RNA aracılı (rehber RNA – Guide RNA – gRNA) nükleazlara dayanır. En yaygın kullanılan sistem, Streptococcus pyogenes’de keşfedilen CRISPR/Cas9 ( CRISPR İlişkili Protein 9 – CRISPR Associated protein 9) sistemidir. CRISPR sistemi bakteri ve arkelerin kendi genomlarını korumaya yönelik bağışıklık sistemlerinin bir parçasıdır. Bu sistem yabancı DNA’yı sekansına bağlı olarak keser, bakteri ve arkeleri istilacı nükleik asitlerden (virüsler gibi) korur.
CRISPR/Cas9 ayrıca bir organizmada birden fazla lokusu hedefleyebilir ve TALEN’ler gibi, bu sistem aynı zamanda diğer işlevlere de uyarlanmıştır. Hatta her hafta yayımlanan bu teknoloji kullanılarak elde edilen yeni belgelerle CRISPR’lar araştırma topluluğuna TALEN’lerden daha kolay ulaşılabilir.
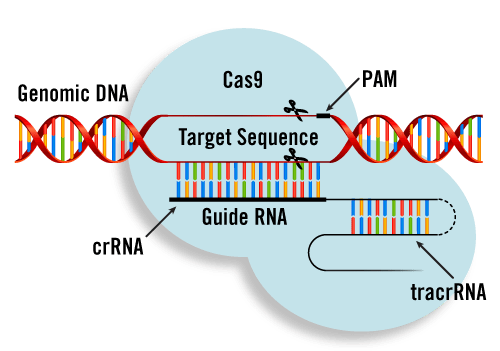
Kaynak : https://www.mobitec.com
Meganükleaz
Meganükleaz teknolojisi ise doğal olarak meydana gelen ana nüklezların DNA bağlanma özgüllüğünün yeniden düzenlenmesiyle ilişkilidir. Meganükleazlar yeniden düzenlenmiş nükleazlar içerisinde en küçük sınıftır. Bu da onları tüm standart gen devretme metotlarına uyumlu hale getirmektedir.
İyi karakterize edilmiş ve genellikle I-CreI ve I-SceI enzimlerini kullananan LAGLIDADG ailesi, ana nükleazların en geniş sınıfıdır. Oranlı tasarım ve seçim kombinasyonları sürecinde bu ana nükleazlar yeni sekansları hedeflemek üzere yeniden düzenlenebilir. Çoğu çalışmada meganükleazlar genom düzenlenme konusunda ümit verirken, ana nükleazların DNA bağlanma ve ayrılma alanlarını birbirinden ayırmanın güç olması, bu yöntemin kullanımını sınırlandırmaktadır. Bu sınırlılıktan dolayı ZFN ve TALEN’lerin yeniden düzenlemede meganükleazlara göre daha avantajlı olduğu belirtilmektedir.
Hedeflenmiş gen düzenlemesi belli bölgeye özgü DNA’yı ayırırken, kalan genomun zarar görmesini önlemeye ve korumaya dayanır. Bu nedenle hedeflenmiş nükleazlar majör odak noktasıdır. 2 zorunlu heterodimer gereksinimiyle fokl dimerizasyonu hedef DNA’ya bağlanmak üzere spesifik oryantasyon ve açılmayı sağlamak üzere ZFN ve TALEN’lerin özgüllüğünü ciddi derecede arttırmaktadır.
ZFN ve TALEN’lerin yapısını andıran, Cas9 nikaz ya da Cas9-fokl füzyonu oluşturmak üzere Cas9 nükleaz alanının inaktivasyonu 2 gRNA/Cas9 kompleksine ihtiyaç duyarak özgüllüğü yükseltmiştir. Ek olarak gRNA ve hedefteki tamamlayıcı alanın 20 nükleotidden 17’ye düşürülmesiyle S. Pyogenes’den elde edilen Cas9’un özgüllüğünü arttırmıştır. Son zamanlarda yapı destekli protein mühendisliği tarafından özgüllüğü arttırılmış yeni Cas9 varyantları üretilmektedir. Tüm bu çalışmalar, CRISPR/Cas nükleazların özgüllüğü hakkındaki başlangıçta var olan endişeleri belirgin bir biçimde azaltmıştır. Bununla birlikte nükleaz teknolojisi gözetilmeksizin, kompleks bir genomdaki hedef ayırmada bütün spektrumu belirlemek zordur. DNA bağlanma özgüllüğü sorusuna ChIP-seq, anlaşılabilir hedef alan tanınması için yeterince bilgilendiricidir. Fakat büyük çoğunluğu hedef olmayan bağlanma bölgeleri nükleaz aktivitelerinin öngöremeyeceği alanlardır.
MAGESTIC
Gen düzenleme sürecini daha kusursuz bir hale getirmek amacıyla yapılan çalışmalar sürmektedir. Joint Institute of Metrology and Biology (Stanford University ve National Institute of Standards and Technology işbirliği) araştırmacıları tarafından yeni bir tür CRISPR platformu olan MAGESTIC (multiplexed accurate genome editing with short, trackable, integrated cellular barcodes – kısa, izlenebilir, bütünleştirilmiş hücresel barkodlarla arttırılmış uygun genom düzenleme) geliştirilmiştir. Bir tür maya olan Saccharomyces cerevisiae’den elde edilen MAGESTIC ile genetik materyalde CRISPR’dan daha hassas ve etkin bir ayrılma sağlanması hedeflenmiştir. Nature Biotechnology’de yapılan yayında MAGESTIC ile gen düzenleme sürecinde hücre sağ kalımının yedi kat arttığı belirtilmektedir.
MAGESTIC’i daha önceki arttırılmış CRISPR düzenlemesi yaklaşımlarından ayıran bir diğer özellik ise yeni bir tür hücresel barkoddur.
Araştırmacılar gelenekselci yaklaşımla plasmid olarak bilinen sirküler DNA’nın küçük parçalarını RNA’ya rehberlik yapması ve her hücrede tasarlanmış mutasyonları izlemek üzere barkodları depolamaları için kullanmıştır. Plasmidler hücre büyüdükçe sayıca artmış ve hücre bölündükçe kalıtılmıştır. Teorik olarak bunlar ödeme standlarındaki siyah beyaz barkodlar gibi çalışmalıdır. Plasmid barkod sayıları her hücrede 10-40 arasında geniş bir aralıkta bulunduğundan hücreler arası zenginlikte uyumsuzluk oluşturmuştur. MAGESTIC’de ise barkodlar bunun yerine kromozomlara entegre edildiğinden daha stabil ve daha sonra bulması ve sayması kolay hale gelmiştir.
Genom düzenleme teknolojilerinin tümü aynı prensiple çalışır. Tek tek genler veya kodlama yapmayan bölgeler için spesifik DNA dizilerini daha sonra dizileri tanıyan, bağlayabilen ve tek iplikli yahut çift iplikli DNA kopmaları oluşturabilen DNA hedefleridir. Örneğin, bir CRISPR “rehber RNA” ile birlikte bir Cas9 proteini, bir hücrenin genomundaki binlerce gen arasında bir hedef gen bulabilir ve hedef bölgedeki her iki DNA şeridini parçalayabilir. Bu, hedef gende bir mutasyon oluşturmak veya düzenlemek için yararlanılabilecek bölünme olayıdır. Hücrenin normal DNA onarım mekanizması daha sonra DNA kopmasını tamir etmeye çalışır. Bu işlemin sonucunda genellikle hedef bölgede bazı DNA dizilerinin silinmesiyle mutasyon oluşur. Ayrı olarak tasarlanmış bir “donör” DNA fragmanı sağlamışsa, onarım mekanizmaları bunu DNA kopmasını düzeltmek için bir şablon olarak kullanabilir ve böylece, tasarlanmış DNA molekülü yeni dizilerin hedef alana girmesine izin verebilir. Bu son işlem birçok potansiyel genom düzenleme uygulamasının anahtarıdır. Çünkü donör DNA fragmanı önceden var olan zararlı mutasyonun veya alternatif olarak yeni, faydalı bir varyantın yerini almak üzere normal bir sekans taşıyabilir. Bu şekilde, hastalığa neden olan mutasyonlar potansiyel olarak düzeltilebilir veya gen fonksiyonunu, hastalığı önleyecek veya tedavi edecek şekilde değiştirmek için yeni mutasyonlar eklenebilir. Böylece, genleri laboratuvarda veya hatta canlı hayvan dokularında düzenlemek hedeflenebilir. Aslında, tasarlanmış nükleazların, birçok memelide olmak üzere çok çeşitli organizmalarda etkili olduğu gösterilmiştir.
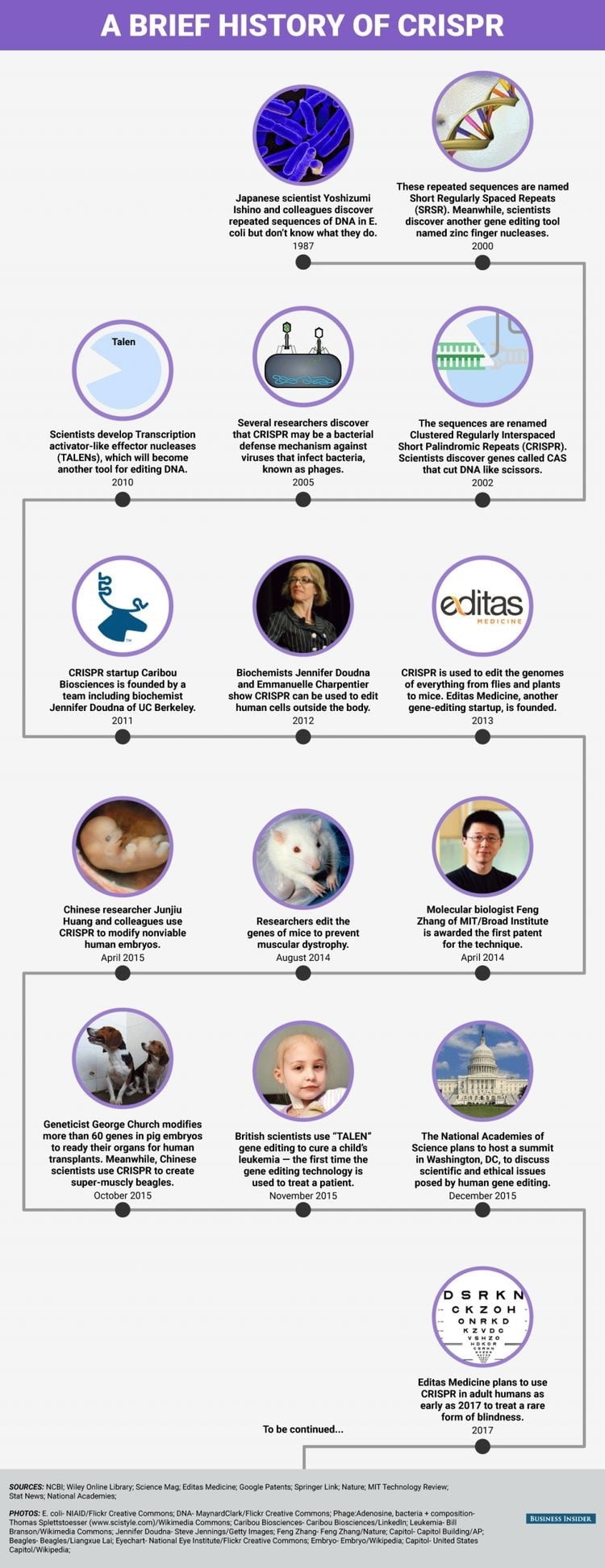
Kaynak : https://www.businessinsider.com
İnsan hücrelerinde de genom düzenlemesi uygulanabilir. HIV enfeksiyonu, orak hücreli anemi ve kanser gibi hastalıklar için hayat kurtarıcı tedaviler sunabilecek hücre bazlı insan terapötiklerinin geliştirilmesinde genom düzenleme araçlarının kullanılmasına büyük ilgi vardır ve bu çalışmalar umut vadetmektedir. MAN2A1-FER olarak adlandırılan bir füzyon geni hedeflenerek ilk gen düzenleme özel olarak kanser füzyon genlerini hedef almak için kullanılmıştır. Ayrıca herediter kanserlerde genom düzenleme çalışmaları devam etmektedir.
CRISPR-Cas9 sistemiyle kistik fibröz hastalarından kültüre edilen hücrelerde CFTR geninin tamiri ile ilgili çalışmalar yapılmıştır. Fare eşey hücrelerinde DNA’yı değiştirerek, dominant katarakt bozukluğu, Tirozinemi tip 1, Duchenne kas distrofi hastalıklarının iyileştirilmesiyle ile ilgili araştırmalar yapılmıştır. Cas9 aynı zamanda viral enfeksiyonların tedavisinde de kullanılma potansiyeline sahiptir ve HIV ve Hepatit B virüsleri için bu sistemin kullanılabilirliği gösterilmiştir. Retinitis pigmentosa, kardiyomiyopatiler gibi patolojiler, kalıtsal kanser, mitokondriyal DNA hastalıkları, nörolojik hastalıklar, böbrek hastalıkları, bağ dokusu hastalıkları, immün yetmezlikler, körlük, sağırlık ve Lynch sendromu, BRCA1 ve BRCA2 genleri ile ilişkili meme ve over kanserleri gibi odaklanabilecek belirli tiple ilişkili kanserler ile ilgili çalışmalar sürmektedir. CRISPR-Cas sistemi ile kazanılan bağışıklığın kalıtsal olarak aktarılması, bu sistemin önemli bir avantajıdır. Genom kurgusunun birçok türün embriyosunda çalışıldığı bilinmektedir.
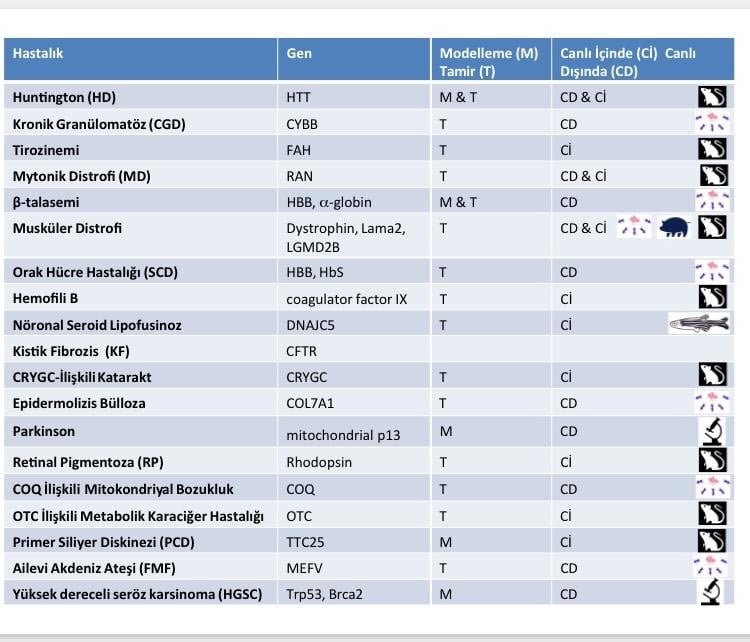
Kaynak : Cihan Taştan
Araştırmacılar, genom düzenleme yöntemlerini kullanarak genlerin model organizmalardaki bitki, hayvan ve enerji hammaddeleri gibi ekonomik açıdan önemli türlerin fonksiyonlarını daha hızlı ve ucuz bir şekilde inceleyebilmektedir. İşlenmiş nükleazların, özellikle CRISPR / Cas9’un, ratlar ve maymunlar gibi memeli embriyolarındaki genleri düzenlemek için kolaylıkla kullanılabileceği gösterilmiştir. Bu embriyolar daha sonra koruyucu hayvanlara implante edilebilir ve DNA’sında değişiklikler taşıyan canlı olarak doğmuş hayvanlar üretilebilir.
Bununla birlikte sonuçta ortaya çıkan hayvanlarda hedef dışı mutagenez, teknolojinin önemli dezavantajları arasında olabilir. İnsan embriyoları ve diğer hayvan embriyoları arasındaki benzerlik, genom düzenleme yöntemlerinin insan destekli üreme prosedürlerine dahil edilebilme olasılığını arttırmaktadır. CRISPR / Cas9’un erken evre insan embriyolarında genleri değiştirmek için kullanılabileceğini gösteren ilk çalışma, 2015 yılı başlarında yayınlanmıştır. Bu deneyler, insan genomlarında açıkça genom düzenlemesinin çalışılabileceğini göstermektedir.
Gen teknolojileri, hayatımıza çok etkin ve hızlı bir şekilde yön vermektedir. Bu doğrultuda yapılan araştırmalar ve araştırmaların sonuçları; ahlaki, yasal, etik açıdan bazı problemleri ve soru işaretlerini de beraberinde getirmektedir. Tartışılan bazı konular; gen kirliliği, doğal dengenin bozulması, genetik bilginin biyolojik silah olarak kullanılabilme riski, sigorta şirketleri tarafından sigorta oranlarının yükseltilmesi, cinsiyet ayrımcılığı, standart sağlık kalitesinin oluşturulamaması, genetik olarak değiştirilmiş ürünlerin insan ve hayvan sağlığı üzerinde olumsuz etkiler oluşturma olasılığı şeklinde sıralanabilir.
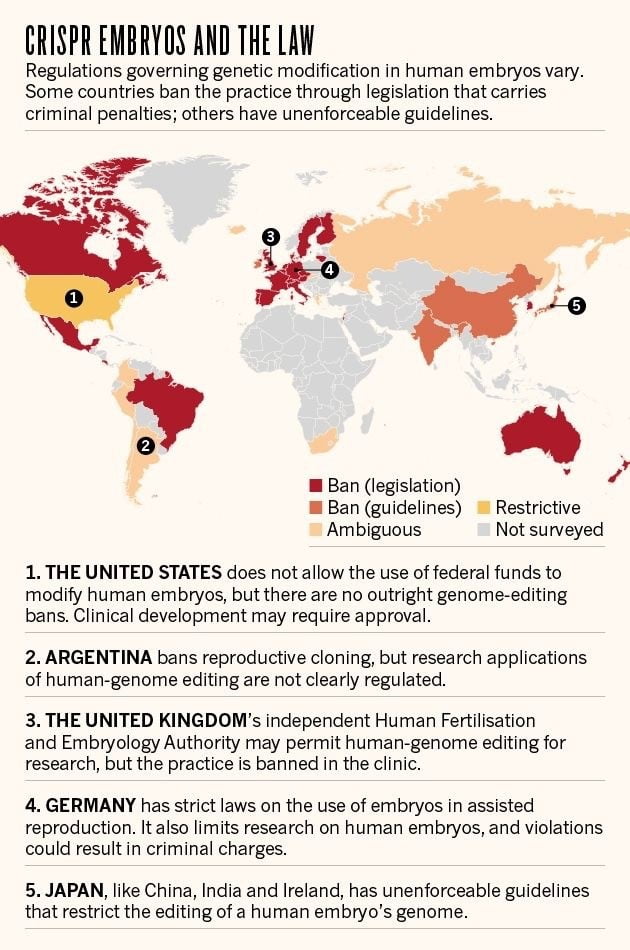
Kaynak : www.nature.com
Embriyo kök hücreleri ile yapılacak çalışmalar şu an tedavisi mümkün olmayan yaygın hastalıklarla mücadelede önemli çözümler sunabilecek bir yöntem olmaya adaydır. Ancak bu çalışmalar, insan embriyolarının bilimsel veya tedavi amaçlı kullanılmasının ve üretilmesinin etik açıdan kabul edilebilir olup olmadığı sorusunu akla getirmektedir. Buna yönelik, ülkeler çeşitli yasal uygulamalar ortaya koymuştur. Örneğin; İrlanda’da araştırma için embriyo kullanmak yasaktır, İspanya tüp bebek girişimleri sırasında fazladan üretilmiş embriyoların ailenin izniyle kullanılmasına izin verirken, tedavi ve araştırma amaçlı embriyo üretilmesi yasaklamıştır, İngiltere’nin ise araştırma, tedavi ve klonlama konularına yaklaşımda en fazla serbestlik sağlayan ülkelerden olduğu söylenebilir.
Teknoloji ilerledikçe doğum öncesi (preimplantasyon) genetik tanı yöntemi daha ayrıntılı ve güvenilir olarak yapılmaktadır. Örneğin; anemili çocuğa sahip kişiler, yeni bir hamilelik öncesi genetik bir ayıklamaya tabi tutulmaktadır. Seçme işlemi sonucu sadece normal embriyoların yaşamasına izin verilerek, doğacak yeni bebekten sağlıklı kemik iliği temin edilmektedir. Bu işlemle ne kadar embriyonun feda edildiği gibi sorulara karşı gene ülkeler farklı politikalar izlemektedir.
Genom düzenleme çalışmalarının gündemde olduğu şu günlerde, Çin’de CRISPR yöntemiyle HIV virüsüne dayanıklı hale getirilen bebeklerin dünyaya gelmesiyle konuya ilişkin tartışmalar artmıştır. Tedavi yönteminin birçok alanda çığır açacak bir teknik olduğu aşikar ancak gelecekte olabilecekler düşünüldüğünde birtakım endişeleri gün yüzüne çıkarmaktadır.
Bahsedilen araştırma konuları ile ilgili gerekli inceleme ve tartışmalar hukukçular, bilim adamları, sağlık personelleri, ahlak bilimciler tarafından kurumsal temelli bir şekilde yapılmaya çalışılmaktadır. Hem bilimsel gelişmelerin önünün gereksiz birtakım endişelerle kesilmesinin engellenmesi hem de insan haklarının, hayvan haklarının korunması ve çevre duyarlılığının canlı tutulması için gerekli gayret gösterilmektedir.
Günümüzde tıp, tarım, hayvancılık, gıda, kimya, enerji ve çevre endüstrileri gibi çok geniş alanlarda gen teknolojilerinden yoğun şekilde yararlanılmaktadır. Bu kullanım alanı teknolojinin ilerlemesi ile daha da artacaktır. Genom düzenlemeleri, hastalık yoktur hasta vardır anlayışının, kişiselleştirilmiş tıbbın ve koruyucu hekimliğin önemini pekiştirecektir. Örneklendirmek gerekirse siz gün gelecek hastalarınıza ya da danışanlarınıza şunları söyleyeceksiniz:
1-Gen dizilemesine bakarak siz de A geni var, bu gen B hastalığına yatkınlık oluşturmaktadır, ilerde B hastalığına yakalanabilirsiniz bunun için genin ekspresyonunu azaltılması gerekmektedir.
2- Sizin hastalığınız C geninden kaynaklanmaktadır ve ilgili çalışmalar yapılacaktır.
3- Sizde D hastalığı var normalde bu hastalar E ilacını kullanır ama sizin gen diziliminiz yapıldı ve sizde E ilacının etkisini azaltan genin olduğu görüldü o yüzden size F ilacı verilecektir. Gibi örnekleri çoğaltabilirsiniz.
Siz; hastanız ya da danışanınız hasta olmadan tedavi edeceksiniz, hastalığın hangi genden kaynaklandığını tespit edeceksiniz, genetik yapısına göre ilaç türünü ve dozunu ayarlayacaksınız.
 Kaynak : http://www.nipple.org.uk/portfolio/what-is-gene-editing-wellcome-rs/
Kaynak : http://www.nipple.org.uk/portfolio/what-is-gene-editing-wellcome-rs/
Hastalıkların genetik olarak temellendirilmesi ve genlerin fonksiyonlarının çözülmesi ile CRİSPR’ın kullanımı artacaktır. CRISPR ile hastaların sağlık durumu, iyilik hali ve yaşam kalitesi artacaktır. Yaşam kalitesinin ve çevre koşullarının düzelmesi ile insanlar daha fazlasını isteyecektir. Daha uzun, daha zeki, daha güçlü, X hastalığa karşı dirençli olmak veya çocuğunda farklı özelliklerin olmasını isteyecektir. Bu da CRISPR’ın preimplantasyon öncesinde kullanım alanında farklılıklara yol açacaktır. Sadece tedavi olmayıp yapay seçilime de olacaktır. Hatta siz yumurtanızı, sperminizi ve paranızı yatırdıktan sonra bebeğinizi istediğiniz özelliklerine göre sipariş edebileceksiniz. Bebek sepeti, genom bank gibi kurumlar kurulabilir. Bu da ciddi etik tartışmalara yol açacaktır. Gen teknolojisinin kullanım alanı etik ve yasal çerçeveye uyumlu olmalıdır.
Şuan güncel olarak tartışılan mitokondriyal bağış, 3 ebeveynli bebek (three parent baby) konusunda da ilerleyen zamanlarda CRISPR’ın mitokondriyal DNA’da kullanılarak hatalı baz ya da genin çıkarılması ile bu etik problemi tamamen ortadan kaldırabilir.
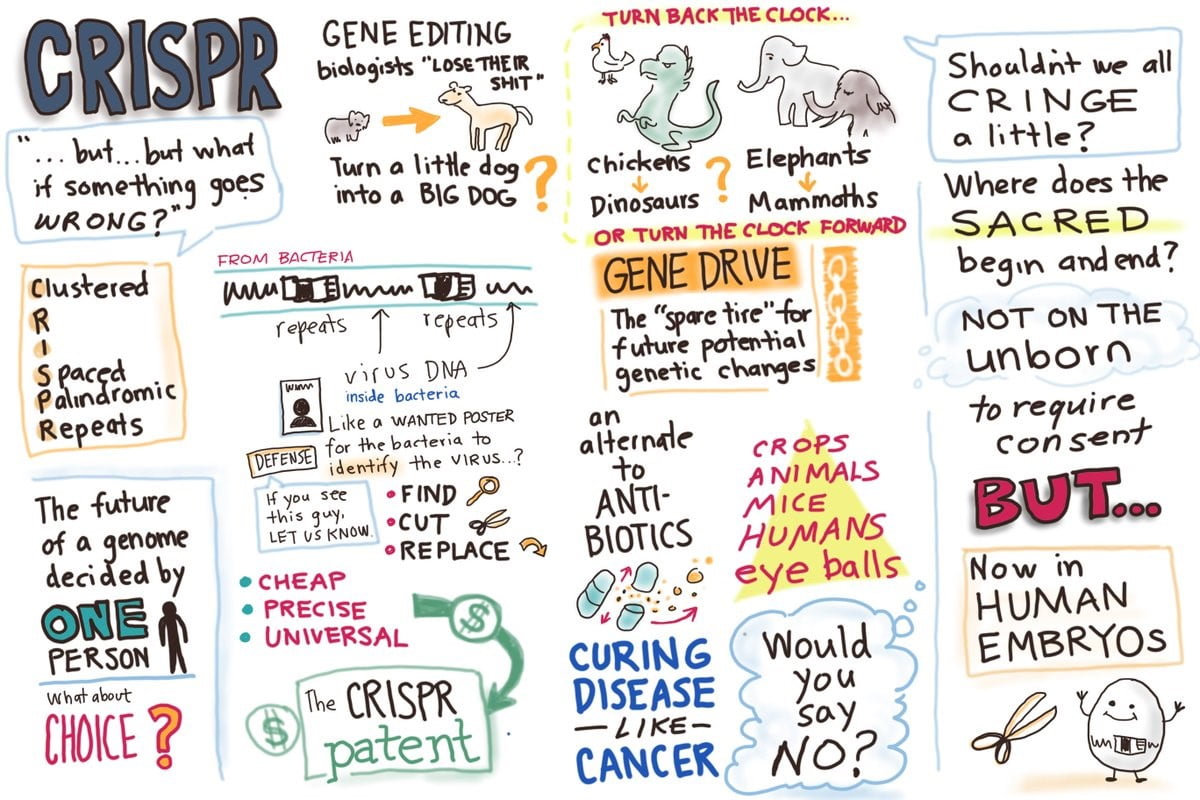
Kaynak : Ward Roberts
Antimikrobiyal olarak da CRISPR teknolojisi kullanılabilir, enfekte olduğunuz bakterinin türüne göre CRISPR antibiyotiği alınabilir ya da hastanelerde sterilizasyonda CRISPR teknolojisi kullanılabilecektir.
CRISPR insan sağlığı için kullanılabildiği gibi bitki ve hayvanlarda da kullanılabilecektir. CRISPR’ın biyoendüstride kullanılmasıyla belki de açlık yok olacaktır. Hayvan ve bitki üretimi geleneksel yapıdan çıkıp biyoteknolojik yöntemlerle üretilecektir. Marketlerde meyve, sebze, et ve süt ürünlerinin satıldığı CRISPR reyonu oluşabilir. Çevre şartlarından dolayı yetiştirilemeyen meyve ve sebzeler her yerde yetiştirilebilecektir. Nesli tükenmekte olan hayvanlarda CRISPR yöntemi kullanılarak nesli tükenmeyen, korunan ve çoğalan hayvanlar üretilebilecektir. Belki de ilerde genetiği değiştirilmiş değil de genetiği seçilmiş bitki ve hayvanların tüketiminden dolayı hastalıklar ortaya çıkacak ve tedavi yöntemleri geliştirilecektir.
Gen teknolojisi, savaşlarda belki de biyolojik silah olarak kullanılacaktır. Tarihin her döneminde insanlığa hizmet etmesi için ortaya konulan buluşlar kötü amaçlar için kullanılmıştır. Ne yazık ki kullanılmaya da devam edecektir. Bu sebepten bilimsel çalışmaların durdurulması veya yasaklanması söz konusu olamaz. Bilimin insanlığın hizmetine sunduğu yenilikler yine bilimsel bir anlayış içinde değerlendirilip temel etik değerlere uygunluğu test edilerek gerekli yasal düzenlemeler yapılmalı ve bu çalışmalar toplum için avantaj haline dönüştürülmelidir.
- Arnould, S., et al., Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. J Mol Biol, 2007. 371(1): p. 49-65.
- Barrangou, R. and L.A. Marraffini, CRISPR-Cas systems: Prokaryotes upgrade to adaptive immunity. Mol Cell, 2014. 54(2): p. 234-44.
- Beurdeley, M., et al., Compact designer TALENs for efficient genome engineering. Nat Commun, 2013. 4: p. 1762.
- Bikard, D., et al., Exploiting CRISPR-Cas nucleases to produce sequence-specific antimicrobials. Nat Biotechnol, 2014. 32(11): p. 1146-50.
- Boissel, S., et al., megaTALs: a rare-cleaving nuclease architecture for therapeutic genome engineering. Nucleic Acids Res, 2014. 42(4): p. 2591-601.
- Bortesi, L. and R. Fischer, The CRISPR/Cas9 system for plant genome editing and beyond. Biotechnol Adv, 2015. 33(1): p. 41-52.
- Chevalier, B.S. and B.L. Stoddard, Homing endonucleases: structural and functional insight into the catalysts of intron/intein mobility. Nucleic Acids Res, 2001. 29(18): p. 3757-74.
- Chevalier, B.S., et al., Design, activity, and structure of a highly specific artificial endonuclease. Mol Cell, 2002. 10(4): p. 895-905.
- Cho, S.W., et al., Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nat Biotechnol, 2013. 31(3): p. 230-2.
- Cong, L., et al., Multiplex genome engineering using CRISPR/Cas systems. Science, 2013. 339(6121): p. 819-23.
- Ding, Q., et al., Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell Stem Cell, 2013. 12(4): p. 393-4.
- Ding, Q., et al., Permanent alteration of PCSK9 with in vivo CRISPR-Cas9 genome editing. Circ Res, 2014. 115(5): p. 488-92.
- Doyon, J.B., et al., Rapid and efficient clathrin-mediated endocytosis revealed in genome-edited mammalian cells. Nat Cell Biol, 2011. 13(3): p. 331-7.
- Dupuy, A., et al., Targeted gene therapy of xeroderma pigmentosum cells using meganuclease and TALEN. PLoS One, 2013. 8(11): p. e78678.
- Robertson, J.A., Ethics and the future of preimplantation genetic diagnosis. Reprod Biomed Online, 2005. 10 Suppl 1: p. 97-101.
- Fu, Y., et al., High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol, 2013. 31(9): p. 822-6.
- Fu, Y., et al., Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol, 2014. 32(3): p. 279-284.
- Gabriel, R., et al., An unbiased genome-wide analysis of zinc-finger nuclease specificity. Nat Biotechnol, 2011. 29(9): p. 816-23.
- Gao, H., et al., Heritable targeted mutagenesis in maize using a designed endonuclease. Plant J, 2010. 61(1): p. 176-87.
- Grizot, S., et al., Efficient targeting of a SCID gene by an engineered single-chain homing endonuclease. Nucleic Acids Res, 2009. 37(16): p. 5405-19.
- Guilinger, J.P., D.B. Thompson, and D.R. Liu, Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification. Nat Biotechnol, 2014. 32(6): p. 577-582.
- Guo, J., T. Gaj, and C.F. Barbas, 3rd, Directed evolution of an enhanced and highly efficient FokI cleavage domain for zinc finger nucleases. J Mol Biol, 2010. 400(1): p. 96-107.
- Gupta, R.M. and K. Musunuru, Expanding the genetic editing tool kit: ZFNs, TALENs, and CRISPR-Cas9. J Clin Invest, 2014. 124(10): p. 4154-61.
- Gurlevik, E., et al., Meganuclease-mediated virus self-cleavage facilitates tumor-specific virus replication. Mol Ther, 2013. 21(9): p. 1738-48.
- Hsu, P.D., E.S. Lander, and F. Zhang, Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014. 157(6): p. 1262-78.
- Hsu, P.D., et al., DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol, 2013. 31(9): p. 827-32.
- Hu, W., et al., RNA-directed gene editing specifically eradicates latent and prevents new HIV-1 infection. Proc Natl Acad Sci U S A, 2014. 111(31): p. 11461-6.
- Wu, Y., et al., Correction of a genetic disease in mouse via use of CRISPR-Cas9. Cell Stem Cell, 2013. 13(6): p. 659-62.
- Hwang, W.Y., et al., Efficient genome editing in zebrafish using a CRISPR-Cas system. Nat Biotechnol, 2013. 31(3): p. 227-9.
- Jinek, M., et al., A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012. 337(6096): p. 816-21.
- Jinek, M., et al., RNA-programmed genome editing in human cells. Elife, 2013. 2: p. e00471.
- Joung, J.K. and J.D. Sander, TALENs: a widely applicable technology for targeted genome editing. Nat Rev Mol Cell Biol, 2013. 14(1): p. 49-55.
- Roy, K.R., et al., Multiplexed precision genome editing with trackable genomic barcodes in yeast. Nat Biotechnol, 2018. 36(6): p. 512-520.
- Kiskinis, E., et al., Pathways disrupted in human ALS motor neurons identified through genetic correction of mutant SOD1. Cell Stem Cell, 2014. 14(6): p. 781-95.
- Kleinstiver, B.P., et al., High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature, 2016. 529(7587): p. 490-5.
- Kleinstiver, B.P., et al., Monomeric site-specific nucleases for genome editing. Proc Natl Acad Sci U S A, 2012. 109(21): p. 8061-6.
- Komor, A.C., A.H. Badran, and D.R. Liu, CRISPR-Based Technologies for the Manipulation of Eukaryotic Genomes. Cell, 2017. 169(3): p. 559.
- Kuscu, C., et al., Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease. Nat Biotechnol, 2014. 32(7): p. 677-83.
- Li, D., et al., Heritable gene targeting in the mouse and rat using a CRISPR-Cas system. Nat Biotechnol, 2013. 31(8): p. 681-3.
- Liang, P., et al., CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell, 2015. 6(5): p. 363-372.
- Lin, S.R., et al., The CRISPR/Cas9 System Facilitates Clearance of the Intrahepatic HBV Templates In Vivo. Mol Ther Nucleic Acids, 2014. 3: p. e186.
- Mali, P., et al., CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering. Nat Biotechnol, 2013. 31(9): p. 833-8.
- Mali, P., et al., RNA-guided human genome engineering via Cas9. Science, 2013. 339(6121): p. 823-6.
- Mandal, P.K., et al., Efficient ablation of genes in human hematopoietic stem and effector cells using CRISPR/Cas9. Cell Stem Cell, 2014. 15(5): p. 643-52.
- Miller, J.C., et al., An improved zinc-finger nuclease architecture for highly specific genome editing. Nat Biotechnol, 2007. 25(7): p. 778-85.
- Niu, Y., et al., Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell, 2014. 156(4): p. 836-43.
- O’Geen, H., et al., A genome-wide analysis of Cas9 binding specificity using ChIP-seq and targeted sequence capture. Nucleic Acids Res, 2015. 43(6): p. 3389-404.
- Ormond, K.E., et al., Human Germline Genome Editing. Am J Hum Genet, 2017. 101(2): p. 167-176.
- Pattanayak, V., et al., High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol, 2013. 31(9): p. 839-43.
- Pattanayak, V., et al., Revealing off-target cleavage specificities of zinc-finger nucleases by in vitro selection. Nat Methods, 2011. 8(9): p. 765-70.
- Pourcel, C., G. Salvignol, and G. Vergnaud, CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology, 2005. 151(Pt 3): p. 653-63.
- Ran, F.A., et al., Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013. 154(6): p. 1380-9.
- Rani, R., et al., CRISPR/Cas9: a promising way to exploit genetic variation in plants. Biotechnol Lett, 2016. 38(12): p. 1991-2006.
- Redondo, P., et al., Molecular basis of xeroderma pigmentosum group C DNA recognition by engineered meganucleases. Nature, 2008. 456(7218): p. 107-11.
- Riviere, J., et al., Variable correction of Artemis deficiency by I-Sce1-meganuclease-assisted homologous recombination in murine hematopoietic stem cells. Gene Ther, 2014. 21(5): p. 529-32.
- Rosen, L.E., et al., Homing endonuclease I-CreI derivatives with novel DNA target specificities. Nucleic Acids Res, 2006. 34(17): p. 4791-800.
- Sander, J.D., et al., In silico abstraction of zinc finger nuclease cleavage profiles reveals an expanded landscape of off-target sites. Nucleic Acids Res, 2013. 41(19): p. e181.
- Seligman, L.M., et al., Mutations altering the cleavage specificity of a homing endonuclease. Nucleic Acids Res, 2002. 30(17): p. 3870-9.
- Singh, P., J.C. Schimenti, and E. Bolcun-Filas, A mouse geneticist’s practical guide to CRISPR applications. Genetics, 2015. 199(1): p. 1-15.
- Slaymaker, I.M., et al., Rationally engineered Cas9 nucleases with improved specificity. Science, 2016. 351(6268): p. 84-8.
- Smith, C., et al., Whole-genome sequencing analysis reveals high specificity of CRISPR/Cas9 and TALEN-based genome editing in human iPSCs. Cell Stem Cell, 2014. 15(1): p. 12-3.
- Smith, J., et al., A combinatorial approach to create artificial homing endonucleases cleaving chosen sequences. Nucleic Acids Res, 2006. 34(22): p. e149.
- Sussman, D., et al., Isolation and characterization of new homing endonuclease specificities at individual target site positions. J Mol Biol, 2004. 342(1): p. 31-41.
- Szczepek, M., et al., Structure-based redesign of the dimerization interface reduces the toxicity of zinc-finger nucleases. Nat Biotechnol, 2007. 25(7): p. 786-93.
- Gyngell, C., T. Douglas, and J. Savulescu, The Ethics of Germline Gene Editing. J Appl Philos, 2017. 34(4): p. 498-513.
- Wyvekens, N., et al., Dimeric CRISPR RNA-Guided FokI-dCas9 Nucleases Directed by Truncated gRNAs for Highly Specific Genome Editing. Hum Gene Ther, 2015. 26(7): p. 425-31.
- Veres, A., et al., Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN targeted human stem cell clones detected by whole-genome sequencing. Cell Stem Cell, 2014. 15(1): p. 27-30.
- Agrotis, A. and R. Ketteler, A new age in functional genomics using CRISPR/Cas9 in arrayed library screening. Front Genet, 2015. 6: p. 300.
- Wolfs, J.M., et al., MegaTevs: single-chain dual nucleases for efficient gene disruption. Nucleic Acids Res, 2014. 42(13): p. 8816-29.
- Nghiem, P.P., et al., Whole genome sequencing reveals a 7 base-pair deletion in DMD exon 42 in a dog with muscular dystrophy. Mamm Genome, 2017. 28(3-4): p. 106-113.
- Wu, X., et al., Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells. Nat Biotechnol, 2014. 32(7): p. 670-6.
- Yen, S.T., et al., Somatic mosaicism and allele complexity induced by CRISPR/Cas9 RNA injections in mouse zygotes. Dev Biol, 2014. 393(1): p. 3-9.
- Yin, H., et al., Genome editing with Cas9 in adult mice corrects a disease mutation and phenotype. Nat Biotechnol, 2014. 32(6): p. 551-3.
- Tazebay, U.H., Genetik Araştırmalar ve Etik. Avrasya Dosyas›, Moleküler Biyoloji ve Gen Teknolojileri Özel, 2002. 8(3): p. 51-61.
- Apak, M., Genetik Hastalıklar ve Prenatal Tanı ile İlgili Etik Sorunlar. Turkiye Klinikleri J Med Ethics, 1994. 2(3): p. 127-130.
- Tauscher, S., Genetik Teknolojisinin Siyasi ve Etik Sınırları: Genetiği Yönetmek. International Journal of Political Studies, 2015. 1.
- Gümüş, S., Genetik ve Etik. Köprü Dergisi, 2003(83).

